[Fonte: Ministero della Salute, documento PDF completo: (LINK). Adattato.]
SORVEGLIANZA VIROLOGICA DELL?INFLUENZA
Approfondimento sui virus A(H1N1)2009 in FASE POST PANDEMICA
agosto 2011
Poich? la stagione 2010/2011 coincide con la fase post pandemica, l?OMS ha raccomandato a tutti i suoi Centri di Collaborazione di condurre studi di approfondimento sia di tipo molecolare sia antigenico sui virus pandemici circolanti nella passata stagione.
In risposta a tale raccomandazione, il NIC-ISS, in collaborazione con alcuni laboratori della rete Influnet, ha condotto analisi filogenetiche, per monitorare l?evoluzione molecolare di questo virus, con particolare riferimento all?HA. Viene qui riportata una breve sintesi di quanto elaborato (dati inviati anche al Centro di Riferimento di Londra).
ANALISI FILOGENETICA DELL?HA DI CEPPI INFLUENZALI A (H1N1) PDM RAPPRESENTATIVI DI QUELLI CIRCOLANTI IN ITALIA NELLA STAGIONE 2010/2011
Le analisi molecolari hanno evidenziato l?emergenza e la simultanea circolazione di 4 distinti gruppi filogenetici (cluster A, B, C e D), che si sono originati a loro volta a partire dall?originario ?clade? 7 (Figura), caratterizzato dalla sostituzione aminoacidica S203T (H1 numbering), rispetto al ceppo A/California/7/2009 (contenuto nel vaccino).
Nella Figura ? riportato il risultato dell?analisi filogenetica effettuata su un totale di 53 ceppi virali, rappresentativi di quelli circolanti in Italia nell?ultima stagione, messi a confronto sia con alcuni isolati della stagione precedente, sia con altri virus isolati in altri Paesi del mondo.
I virus appartenenti al cluster A risultano essere accomunati da 3 mutazioni aminoacidiche, R205K, I216V e V249L, a cui si aggiunge una quarta interessante sostituzione nel sito antigenico Ca2, H138Q, presente in un sottogruppo di ceppi virali afferenti a questo cluster.
Il cluster B comprende, invece, virus caratterizzati dalla mutazione N125D (sito antigenico Sa), gi? riportata negli isolati dell?Emisfero Sud (1) gi? all?inizio del 2010.
Nel cluster C si raggruppa un consistente numero di isolati virali, accomunati dalla sostituzione S185T (sito antigenico Sb), questo gruppo risulta essere quello dominante e in forte espansione a livello mondiale (2,3). Anche la sostituzione D97N risulta spesso associata ai virus appartenenti a questo cluster, mentre le mutazioni S143G e A197T definiscono un distinto sottogruppo filogenetico.
Il cluster D, a cui afferisce il minor numero di sequenze virali italiane qui analizzate, include anche sequenze di virus isolati di recente in UK (Ellis et al. 2011) e caratterizzati dalle mutazioni A134T e S183P.
Nessuno dei virus A(H1N1) appartenenti ai suddetti gruppi genetici ha comunque mostrato cambiamenti antigenici significativi rispetto al ceppo vaccinale A/California/7/2009, come mostrato nella tabella, in cui sono riportati i risultati dell?analisi sierologica (test HI), condotta su alcuni isolati. Detti isolati sono da considerarsi rappresentativi del totale dei virus circolanti.

Figura - Relazioni filogenetiche relative al dominio HA1 della HA di recenti isolati umani A(H1N1) pandemici in Italia. L?analisi filogenetica ? stata eseguita utilizzando il metodo Maximum Likelihood per il calcolo delle distanze genetiche. Le sequenze incluse nell?albero sono state ottenute in ISS o fornite dai laboratori della rete (■). Tutte le sequenze di riferimento (in rosso) sono state ottenute da NCBI Influenza Virus Resource (http://www.ncbi.nlm.nih.gov/genomes/FLU/) e sono rappresentative dei virus circolanti nel mondo nel 2010. Le sequenze dei virus italiani isolati da casi gravi nella stagione 2010/2011 sono indicate in grassetto; il simbolo (?) indica le sequenze dei virus isolati dai casi fatali. I virus isolati nella stagione precedente (2009/2010) in Italia sono evidenziate dal simbolo (▲). I virus con mutazioni rilevate nel residuo aminoacidico 222 sono segnalati nell?albero. Le principali mutazioni aminoacidiche (H1 numbering) sono riportate sui nodi dell?albero.
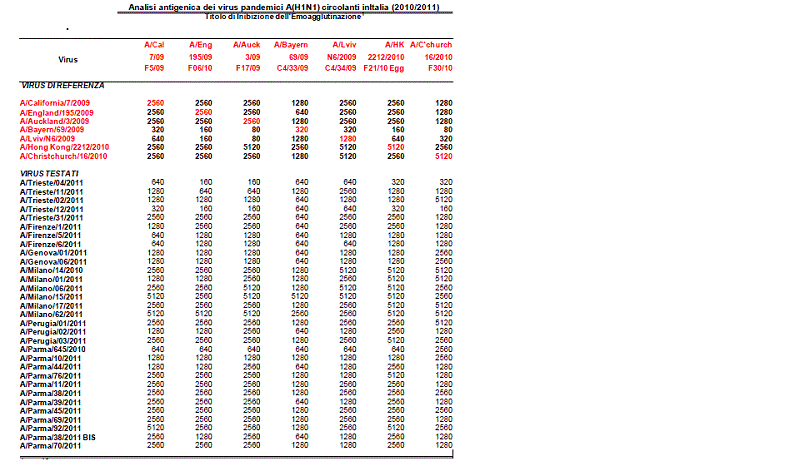
Tabella - I risultati delle caratterizzazioni antigeniche hanno mostrato, nella maggioranza dei casi, una quasi totale correlazione antigenica con il ceppo vaccinale 2010-2011 (A/California/7/2009). Tuttavia un numero limitato di ceppi ha mostrato una attivit? lievemente ridotta verso il ceppo vaccinale e varianti antigenicamente indistinguibili come il ceppo A/Christchurch/16/2010, dovuta a cambiamenti aminoacidici tra le posizioni 153-157 della HA,.
Bibliografia:
1. Barr IG, Cui L, Komadina N, et al. A new pandemic influenza A(H1N1) genetic variant predominated in the winter 2010 influenza season in Australia, New Zealand and Singapore. Euro Surveill. 2010;15(42):pii=19692.
2. Ellis J, Galiano M, Pebody R, et al. Virological analysis of fatal influenza cases in the United Kingdom during the early wave of influenza in winter 2010/11. Euro Surveill. 2011;16(1):pii=19760.
3. WHO Influenza Report prepared for the WHO annual consultation on the composition of influenza vaccine for the Northern Hemisphere (14th - 17th February 2011)- WHO Influenza Centre, London, UK (LINK)
-
-------
SORVEGLIANZA VIROLOGICA DELL?INFLUENZA
Approfondimento sui virus A(H1N1)2009 in FASE POST PANDEMICA
agosto 2011
Poich? la stagione 2010/2011 coincide con la fase post pandemica, l?OMS ha raccomandato a tutti i suoi Centri di Collaborazione di condurre studi di approfondimento sia di tipo molecolare sia antigenico sui virus pandemici circolanti nella passata stagione.
In risposta a tale raccomandazione, il NIC-ISS, in collaborazione con alcuni laboratori della rete Influnet, ha condotto analisi filogenetiche, per monitorare l?evoluzione molecolare di questo virus, con particolare riferimento all?HA. Viene qui riportata una breve sintesi di quanto elaborato (dati inviati anche al Centro di Riferimento di Londra).
ANALISI FILOGENETICA DELL?HA DI CEPPI INFLUENZALI A (H1N1) PDM RAPPRESENTATIVI DI QUELLI CIRCOLANTI IN ITALIA NELLA STAGIONE 2010/2011
Le analisi molecolari hanno evidenziato l?emergenza e la simultanea circolazione di 4 distinti gruppi filogenetici (cluster A, B, C e D), che si sono originati a loro volta a partire dall?originario ?clade? 7 (Figura), caratterizzato dalla sostituzione aminoacidica S203T (H1 numbering), rispetto al ceppo A/California/7/2009 (contenuto nel vaccino).
Nella Figura ? riportato il risultato dell?analisi filogenetica effettuata su un totale di 53 ceppi virali, rappresentativi di quelli circolanti in Italia nell?ultima stagione, messi a confronto sia con alcuni isolati della stagione precedente, sia con altri virus isolati in altri Paesi del mondo.
I virus appartenenti al cluster A risultano essere accomunati da 3 mutazioni aminoacidiche, R205K, I216V e V249L, a cui si aggiunge una quarta interessante sostituzione nel sito antigenico Ca2, H138Q, presente in un sottogruppo di ceppi virali afferenti a questo cluster.
Il cluster B comprende, invece, virus caratterizzati dalla mutazione N125D (sito antigenico Sa), gi? riportata negli isolati dell?Emisfero Sud (1) gi? all?inizio del 2010.
Nel cluster C si raggruppa un consistente numero di isolati virali, accomunati dalla sostituzione S185T (sito antigenico Sb), questo gruppo risulta essere quello dominante e in forte espansione a livello mondiale (2,3). Anche la sostituzione D97N risulta spesso associata ai virus appartenenti a questo cluster, mentre le mutazioni S143G e A197T definiscono un distinto sottogruppo filogenetico.
Il cluster D, a cui afferisce il minor numero di sequenze virali italiane qui analizzate, include anche sequenze di virus isolati di recente in UK (Ellis et al. 2011) e caratterizzati dalle mutazioni A134T e S183P.
Nessuno dei virus A(H1N1) appartenenti ai suddetti gruppi genetici ha comunque mostrato cambiamenti antigenici significativi rispetto al ceppo vaccinale A/California/7/2009, come mostrato nella tabella, in cui sono riportati i risultati dell?analisi sierologica (test HI), condotta su alcuni isolati. Detti isolati sono da considerarsi rappresentativi del totale dei virus circolanti.
Figura - Relazioni filogenetiche relative al dominio HA1 della HA di recenti isolati umani A(H1N1) pandemici in Italia. L?analisi filogenetica ? stata eseguita utilizzando il metodo Maximum Likelihood per il calcolo delle distanze genetiche. Le sequenze incluse nell?albero sono state ottenute in ISS o fornite dai laboratori della rete (■). Tutte le sequenze di riferimento (in rosso) sono state ottenute da NCBI Influenza Virus Resource (http://www.ncbi.nlm.nih.gov/genomes/FLU/) e sono rappresentative dei virus circolanti nel mondo nel 2010. Le sequenze dei virus italiani isolati da casi gravi nella stagione 2010/2011 sono indicate in grassetto; il simbolo (?) indica le sequenze dei virus isolati dai casi fatali. I virus isolati nella stagione precedente (2009/2010) in Italia sono evidenziate dal simbolo (▲). I virus con mutazioni rilevate nel residuo aminoacidico 222 sono segnalati nell?albero. Le principali mutazioni aminoacidiche (H1 numbering) sono riportate sui nodi dell?albero.
Tabella - I risultati delle caratterizzazioni antigeniche hanno mostrato, nella maggioranza dei casi, una quasi totale correlazione antigenica con il ceppo vaccinale 2010-2011 (A/California/7/2009). Tuttavia un numero limitato di ceppi ha mostrato una attivit? lievemente ridotta verso il ceppo vaccinale e varianti antigenicamente indistinguibili come il ceppo A/Christchurch/16/2010, dovuta a cambiamenti aminoacidici tra le posizioni 153-157 della HA,.
Bibliografia:
1. Barr IG, Cui L, Komadina N, et al. A new pandemic influenza A(H1N1) genetic variant predominated in the winter 2010 influenza season in Australia, New Zealand and Singapore. Euro Surveill. 2010;15(42):pii=19692.
2. Ellis J, Galiano M, Pebody R, et al. Virological analysis of fatal influenza cases in the United Kingdom during the early wave of influenza in winter 2010/11. Euro Surveill. 2011;16(1):pii=19760.
3. WHO Influenza Report prepared for the WHO annual consultation on the composition of influenza vaccine for the Northern Hemisphere (14th - 17th February 2011)- WHO Influenza Centre, London, UK (LINK)
-
-------